Introduzione
Il metabolismo cellulare è l’insieme coordinato delle reazioni biochimiche che consentono alle cellule di ottenere energia, sintetizzare componenti e mantenere l’omeostasi. Fondamento della fisiologia cellulare, è strettamente regolato sia da stimoli interni che esterni, con conseguenti ripercussioni sullo stato e salute dell’intero organismo.
Numerosi attori giocano un ruolo nel metabolismo cellulare, sia a livello di strutture cellulari che di composti molecolari (Figura 1). Gli organelli coinvolti sono svariati, ma il protagonista principale è senza dubbio il network mitocondriale. Celebre struttura sub-cellulare, il mitocondrio rappresenta la centrale elettrica della cellula, essendo il principale deputato alla produzione di energia (AppNoteMitochondria). Coinvolti anche nell’omeostasi del calcio e nella regolazione dello stress ossidativo, i mitocondri producono grandi quantità di ATP (Adenosintrifosfato) attraverso un processo denominato fosforilazione ossidativa. L’ATP è considerato la moneta energetica della cellula e rappresenta la fonte di energia principale; questa molecola viene continuamente rigenerata attraverso due vie metaboliche principali: la glicolisi e la fosforilazione ossidativa. Questi due processi catabolici sono finemente regolati e determinano lo stato metabolico della cellula. Un ruolo cruciale in queste due vie metaboliche è ricoperto dal NADH, un coenzima che esiste in forma libera (citoplasmatica) e legata a proteine (soprattutto mitocondriali) in base allo stato metabolico della cellula.
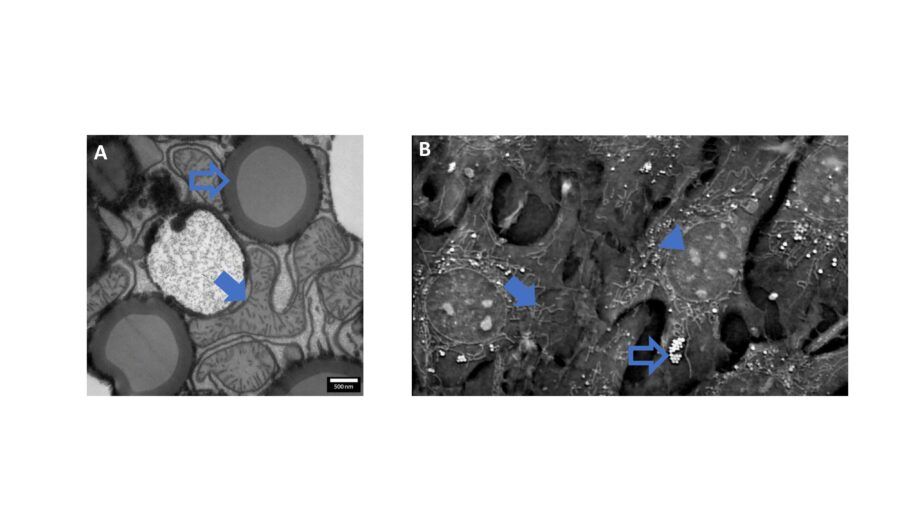
Figura 1: A. Sezione di tessuto nella quale si può apprezzare la struttura dei mitocondri (freccia) e delle lipid droplets (freccia vuota). Questa immagine è stata ottenuta con il microscopio a trasmissione (TEM) LVEM25 e concessa da Delong Instruments. B. Immagine olotomografica ottenuta con Nanolive 3D Cell Explorer. In questo caso è possibile osservare mitocondri (freccia), lipid droplets (freccia vuota) e nucleo (freccia corta). A differenza dell’immagine ottenuta al TEM, l’immagine olotomografica viene acquisita su campioni cellulari vivi, mantenendo comunque un’ottima risoluzione.
Ad attirare particolare interesse negli ultimi decenni sono le Lipid Droplets (LDs), organelli poco considerati nel secolo scorso a causa della loro natura apparentemente inerte, ma che in realtà si stanno rivelando player importantissimi nel metabolismo lipidico. Come trattato nella nostra precedente Application Note (AppNoteLipidDroplets), le LDs sono infatti coinvolte nell’immagazzinamento e gestione di svariate tipologie di lipidi, ma giocano un ruolo essenziale interagendo con altri organelli come mitocondri, perossisomi, reticolo endoplasmatico e lisosomi, orchestrando il rilascio e il riutilizzo degli acidi grassi in base allo stato metabolico cellulare.
A completare il complesso quadro del metabolismo cellulare vi sono vari organelli come il reticolo endoplasmatico, l’apparato di Golgi, i perossisomi, i lisosomi ecc. oltre a numerose reazioni biochimiche. Il metabolismo cellulare è dunque estremamente complesso e finemente regolato a più livelli. Alterazioni del metabolismo spesso si traducono in gravi patologie come tumori, diabete o malattie neurodegenerative; di conseguenza, studiare e determinare lo stato metabolico delle cellule e dei tessuti diventa essenziale sia in fase diagnostica che terapeutica. Numerosi laboratori concentrano i propri studi proprio sul metabolismo cellulare, cercando di comprendere i meccanismi cellulari che lo regolano e ricercando nuove strategie terapeutiche. Tra le tecniche utilizzate per l’analisi del metabolismo, la microscopia è una delle più utilizzate in quanto consente di visualizzare lo stato di vari organelli e molecole metaboliche. Principalmente, la tecnica più utilizzata è la microscopia a fluorescenza, tuttavia negli ultimi anni una crescente porzione di ricercatori predilige l’utilizzo di tecniche label-free, ovvero strategie di imaging dove marcatori e staining non sono necessari. Questa tipologia di imaging consente di eliminare eventuali artefatti e soprattutto bias dovuti alla fototossicità intrinseca alla fluorescenza. In questa application note andremo a descrivere 3 strategie di imaging label-free dedicate allo studio del metabolismo cellulare.
L’olotomografia per visualizzare Lipid Droplets senza fluorescenza
Le Lipid Droplets (LD) sono state considerate per lungo tempo serbatoi passivi di lipidi all’interno delle cellule eucariotiche. Ignorate per quasi un secolo, recenti studi hanno rivelato che in realtà sono organelli molto dinamici e che giocano un ruolo essenziale nel metabolismo cellulare. Dagli anni 90 infatti, numerosi studi hanno attribuito svariate funzioni alle LD, come regolazione del metabolismo lipidico, stress cellulare, membrane trafficking e trasduzione di segnali. Inoltre, la loro associazione con importanti patologie come cancro, obesità e diabete ha generato grande interesse da parte di tutta la comunità scientifica.
La loro visualizzazione è relativamente semplice: disponendo di numerosi marcatori e avendo dimensioni relativamente grandi le LD vengono comunemente visualizzate utilizzando la microscopia a fluorescenza. Tuttavia, essendo organelli estremamente dinamici, la loro visualizzazione nel tempo diventa essenziale al fine di monitorare i loro cambiamenti e interazioni in tempo reale. Mentre la microscopia elettronica e a fluorescenza offrono solamente immagini istantanee del campione, gli esperimenti di live-imaging possono fornire informazioni nel tempo. Come menzionato però, l’utilizzo della fluorescenza negli esperimenti di live-imaging può rivelarsi sfavorevole e introdurre bias indesiderati. L’olotomografia di Nanolive è una tecnica che permette di ottenere time-lapse di colture cellulari senza l’utilizzo di marcatori a fluorescenza, proponendosi dunque come una tecnica perfetta per l’osservazione delle lipid droplets. Infatti, l’olotomografia consiste nella quantificazione diretta dell’indice di rifrazione e fornisce immagini ad alta risoluzione. Nello specifico, le LD, avendo un indice di rifrazione ben specifico, risultano evidenti nei video Nanolive (Figura 2A). Di conseguenza, non solo la loro visualizzazione diventa immediata, ma anche la loro analisi è estremamente semplice. Nanolive infatti, propone un software dedicato in grado di riconoscere e quantificare tutte le lipid droplets presenti nei video olotomografici (Figura 2B). Grazie all’intelligenza artificiale, il module d’analisi Smart Lipid Droplet Assay (SLDA), segmenta le LD e ne quantifica numerosi parametri: dalla morfologia al contenuto, dal numero alla distribuzione all’interno della cellula, SLDA fornisce con un solo click una descrizione dettagliatissima delle LD nel tempo.
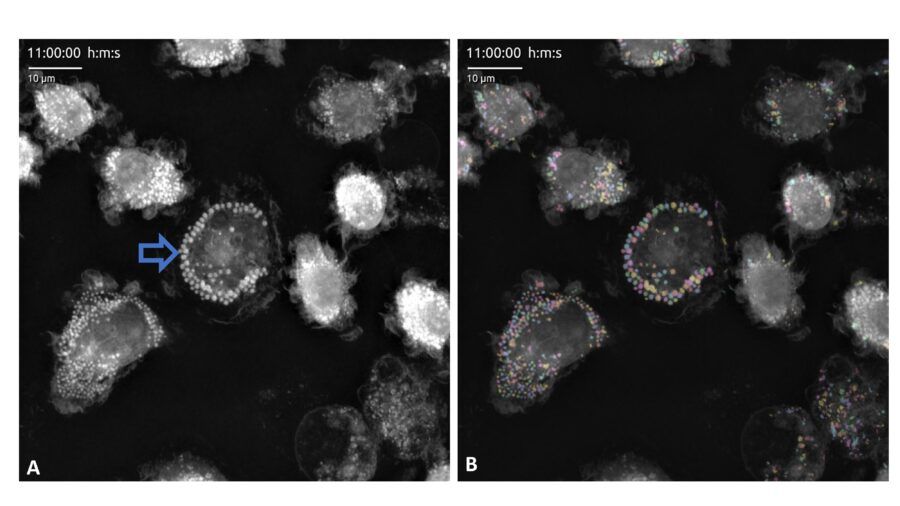
Figura 2: A. Frame di un video olotomografico ottenuto con Nanolive 3D Cell Explorer, nel quale numerose LD appaiono attorno al nucleo delle cellule presenti (freccia vuota). Grazie al loro indice di rifrazione specifico, le LD risultano evidenti e ben definite. B. Maschera generata da SLDA, modulo d’analisi in grado di segmentare le LD. In questo frame è possibile apprezzare l’altissima efficienza del software, il quale riconosce tutte le LD presenti senza errori.
Un esempio di come SLDA possa velocizzare il workflow di un’analisi di LD è riportato in figura 3, nella quale si possono osservare 5 frame di un video olotomografico di 8 ore. Nello specifico, l’esperimento mostra l’accumulo di LD in pre-adipociti dopo la somministrazione di acido oleico, composto in grado di stimolare la formazione di LD. Il modulo d’analisi SLDA riconosce sia le cellule che le LD nel tempo, andando a quantificare svariati parametri: alcune metriche principali sono riportate in figura 3B. SLDA fornisce una dettagliata descrizione delle LD, quantificando il loro accumulo numericamente (conta assoluta o per cellula), ma anche a livello di dimensioni (area, um2) e contenuto (massa secca, pg). Inoltre, è in grado di determinare la posizione media delle LD rispetto al centroide della cellula “parentale” (distribuzione, um).

Figura 3: A. 5 frame di un video ottenuto con Nanolive 3D Cell Explorer di 8 ore. La prima riga mostra i frame del video olotomografico, mentre le righe seguenti mostrano le maschere generate da SLDA; cellule, Lipid Droplets e Cellule/Lipid Droplets. B. 6 esempi di dati ottenibili con SLDA. Il software è in grado di quantificare con un solo click numerose metriche, fornendo una descrizione completa dell’intera popolazione cellulare e le sue LD. Dalla conta a dati morfologici come area, volume e contenuto, SLDA semplifica l’analisi delle LD senza l’utilizzo di marcatori a fluorescenza.
Visualizzare i mitocondri senza marcatori? Con Nanolive si può!
Quando si tratta di metabolismo cellulare, il protagonista principale è senza dubbio il mitocondrio. All’interno di questo organello avvengono importanti reazioni cataboliche come il ciclo di Krebs, la catena di trasporto degli elettroni e la fosforilazione ossidativa (nel complesso detta respirazione mitocondriale), ma anche processi anabolici come la sintesi di amminoacidi o intermedi metabolici. Lo studio dei mitocondri è estremamente diffuso e importante in svariati campi di ricerca. La loro visualizzazione è evoluta nel tempo, partendo dalla microscopia elettronica a trasmissione fino ad arrivare alla microscopia a super-risoluzione. Attualmente, numerose strategie vengono applicate per visualizzare e analizzare il network mitocondriale, ma, come per le LD, esperimenti in time-lapse risultano molto informativi e utili per organelli particolarmente dinamici. Anche in questo caso, l’olotomografia di Nanolive si presta perfettamente a questo scopo, in quanto consente di visualizzare i mitocondri in label-free e nel tempo, grazie al loro indice di rifrazione specifico.
I mitocondri sono organelli estremamente dinamici e nei video olotomografici di Nanolive si percepisce subito la loro motilità. Il network mitocondriale è infatti in continuo movimento e costante rimodellamento, con fusioni e divisioni degli stessi organelli. Visualizzare tutti questi eventi è un’impresa non indifferente e fortunatamente l’olotomografia di Nanolive consente di registrarli senza necessità di colorazioni fluorescenti e senza danneggiare il campione. Una volta ottenuto il video, l’impresa non è compiuta: quantificare tutti questi cambiamenti è essenziale al fine di poter determinare lo stato metabolico cellulare. Fortunatamente Nanolive ha rilasciato recentemente un software in grado di riconoscere e quantificare i mitocondri. Lo Smart Mitochondrial Assay (SMtA) è un modulo d’analisi dedicato allo studio dei mitocondri; grazie all’intelligenza artificiale allenata negli ultimi anni con migliaia di terabyte di dati, Nanolive è ora in grado di fornire una soluzione efficiente per la segmentazione dei mitocondri in svariate tipologie cellulari (Figura 4). Dallo studio della loro forma, dimensione e contenuto, fino alla quantificazione dei “branches”, terminazioni e giunzioni tra mitocondri: SMtA permette di analizzare con grande precisione dinamiche mitocondriali fondamentali come fissione, fusione, “swelling”, mitofagia e complessità del network, cruciali per valutarne l’attività metabolica.

Figura 4: Frame di un video olotomografico ottenuto con Nanolive 3D Cell Explorer, nel quale i mitocondri sono stati segmentati utilizzando SMtA. Il network mitocondriale appare infatti “colorato” a dimostrazione dell’efficiente riconoscimento avvenuto tramite intelligenza artificiale.
A riprova delle potenzialità di SMtA, in figura 5 è possibile apprezzare un esperimento effettuato e analizzato con Nanolive. Nello specifico, una coltura cellulare è stata trattata con un chemioterapico (AntimycinA), il cui meccanismo agisce colpendo il network mitocondriale (per ulteriori dettagli, leggere l’articolo NanoliveAntimycinA). Dopo aver registrato un video in time-lapse per 30 ore (frequenza 16 minuti) della coltura cellulare trattata e controllo, i due video sono stati analizzati con SMtA per valutare lo stato dei mitocondri (Figura 5A). In figura 5B è possibile osservare 3 grafici dove il numero di ramificazioni (branches), terminazioni (endpoints) e giunzioni (junctions) mitocondriali sono riportati: mentre nella popolazione di controllo il network mitocondriale aumenta la sua estensione, le cellule del campione trattato riducono il loro network confermando l’azione nociva a livello mitocondriale del composto.

Figura 5: A. Frame di due video olotomografici di due colture cellulari, una trattata con Antimycin A (riquadro grande) e una controllo (riquadro piccolo). B. 3 esempi di grafici ottenuti con SMtA. Nello specifico, viene mostrata la quantificazione del numero di ramificazioni, terminazioni e giunzioni mitocondriali: in verde i dati relativi al controllo, in arancione quelli del trattato.
Determinare lo stato metabolico delle cellule sfruttando l’autofluorescenza
Nelle sezioni precedenti abbiamo compreso l’importanza del metabolismo cellulare: dai meccanismi della biologia cellulare fino allo studio di malattie, determinare lo stato metabolico di una cellula è fondamentale per comprenderne la sua funzione, salute e identità. A giocare un ruolo centrale nel metabolismo è il NADH, molecola chiave per la produzione di energia nelle cellule. In base alla sua forma (libera o legata a proteine mitocondriali) il NADH ci racconta quale via metabolica sta utilizzando la cellula: glicolisi (produzione rapida ma meno efficace), oppure fosforilazione ossidativa (produzione più stabile e duratura). Di conseguenza, per comprendere lo stato metabolico della cellula è sufficiente determinare quale forma di NADH prevale all’interno della cellula: cellule che presentano più NADH libero, saranno da considerarsi glicolitiche, mentre cellule con una maggior frazione di NADH legato, fosforilative.
Una strategia di imaging in grado di determinare la presenza di legami tra proteine è la Fluorescence Lifetime Imaging Microscopy (FLIM). Come descritto nella nostra precedente Application Note (Fluorescence Lifetime Analysis), questo approccio sfrutta il tempo di decadimento del fluoroforo e consente di discriminare con alta precisione variazioni nello stato conformazionale delle molecole. È interessante notare che il NADH è una molecola autofluorescente e che la sua “lifetime signature” (tempo di decadimento specifico) varia notevolmente tra le sue due forme libera e legata. Di conseguenza ne deriva che attraverso l’implementazione di sistemi FLIM è possibile determinarne lo stato del NADH e dunque l’attività metabolica cellulare. Sfruttando l’autofluorescenza della molecola d’interesse, questo approccio non necessita l’utilizzo di alcun marcatore o sonda fluorescente; determinare lo stato metabolico delle cellule attraverso la FLIM è dunque una tecnica label-free, non invasiva, rapida e riproducibile.
Nello specifico, il NADH è una molecola che assorbe nel range da 300 a 400nm (con picco di assorbimento a 340nm). Eccitare il NADH è possibile sia con sorgenti laser nel campo ultravioletto (UV) che nel vicino infrarosso (NIR). Nella microscopia a due fotoni vengono utilizzati laser con lunghezze d’onda maggiori (NIR) e ciò introduce due grandi vantaggi: minor fototossicità e maggior penetrazione del campione. Queste caratteristiche rendono la microscopia multifotone adatta all’osservazione di campioni vivi e tridimensionali. Ne consegue che integrando la FLIM alla microscopia due fotoni otterremo uno strumento perfetto per determinare l’attività metabolica di cellule in campioni complessi e in live-imaging.

Figura 6: A. Immagine FLIM di una sezione di tessuto polmonare. Ogni pixel dell’immagine viene classificato in base al tempo di arrivo medio dei fotoni: colori più caldi indicano Fluorescence Lifetime veloci (circa 0.4 ns) e colori più freddi tempistiche più lente (circa 3.4 ns). Grazie all’analisi dei fasori fornita dal software FLIM LABS, è possibile ottenere un Phasor-Plot dove ogni pixel dell’immagine è posizionato nel grafico in base al suo Lifetime. Ciò consente una visualizzazione rapida e intuitiva della “Nuvola” o “Fluorescence Lifetime Signature”. B. Metabolic Imaging con FLIM LABS. L’immagine FLIM funziona come una vera e propria mappa metabolica, in cui ogni pixel è codificato con un colore che riflette l’attività metabolica di quel punto nel campione: fosforilativa o glicolitica. L’assegnazione avviene attraverso l’analisi del phasor plot lungo la Metabolic Trajectory: ogni punto dell’immagine viene classificato in base alla vicinanza alla Fluorescence Lifetime associata alla glicolisi (0,4 ns) o alla fosforilazione ossidativa (3,4 ns). Definendo un gradiente di colore è possibile generare una colormap che varia dal blu chiaro per la glicolisi al viola per la fosforilazione ossidativa, consentendo una lettura visiva immediata dello stato energetico delle cellule.
In figura 6 è possibile osservare un esempio di Metabolic Imaging effettuato con i dispositivi FLIM LABS, azienda specializzata nella produzione di device per Fluorescence Lifetime Analysis (FLA). Nello specifico, la configurazione utilizzata consisteva in un microscopio due fotoni Bruker ULTIMA integrato con una scheda di acquisizione FLIM LABS. Grazie a questo set-up è stato possibile determinare il tempo di decadimento del NADH, stimolando la molecola con il laser a 780nm e registrando il tempo di arrivo dei fotoni con la FLIM LABS Data Acquisition Card (Figura 6A). Nello specifico, è stato determinato il tempo di decadimento del NADH libero (0.4ns) e del NADH legato (3.4ns), per poi visualizzarli sfruttando l’analisi dei fasori (Phasor-Plot). Grazie ai software d’analisi FLIM LABS è infatti possibile graficare i dati di Fluorescence Lifetime con i Phasor-Plot, diagrammi in cui ogni pixel dell’immagine viene trasformato in un punto sul grafico in base al suo “Lifetime” (Figura 6A). Rappresentando i dati in questo modo è possibile visualizzare rapidamente e intuitivamente le “posizioni” dei tempi di arrivo dei fotoni e dunque le posizioni caratteristiche delle due forme di NADH. Collegando le due posizioni otteniamo la Metabolic Trajectory e ciò consente di visualizzare la distribuzione e il bilanciamento tra NADH libero e legato, restituendo così una vera e propria mappa metabolica del tessuto o della cellula analizzata (Figura 6B). In parole povere, se il Phasor-plot sarà caratterizzato da una “nuvola” di punti più spostata verso 3.4 ns, il campione sarà caratterizzato da una frazione di NADH legato maggiore (dunque un’attività metabolica fosforilativa/ossidativa), mentre se la “nuvola” sarà localizzata verso l’altro lato della Metabolic Trajectory il campione sarà caratterizzato da una popolazione di NADH libero maggiore (dunque un’attività metabolica glicolitica).

Figura 7: Immagine FLIM di uno sferoide tumorale. Oltre all’immagine “classica” (bianco e nero) dove registriamo l’intensità della fluorescenza, grazie a FLIM LABS è stato possibile determinare lo stato metabolico delle cellule all’interno dello sferoide. Utilizzando la microscopia a 2 fotoni è stato possibile visualizzare anche gli strati interni dell’aggregato cellulare tridimensionale, penetrando il campione senza danneggiarlo in real-time. Nonostante siano stati assegnati due colorazioni differenti, la totalità delle cellule dello sferoide in analisi presentano un metabolismo ossidativo; nel Phasor-Plot è possibile notare come la “nuvola” è localizzata completamente a sinistra (3.4ns).
Un ulteriore esempio lo ritroviamo in figura 7 dove in questo caso, sempre con lo stesso set-up (FLIM LABS integrato su Bruker ULTIMA), è stato quantificato il metabolic index di uno sferoide. Gli sferoidi sono aggregati cellulari tridimensionali derivanti da una singola tipologia cellulare. In questo esempio, la coltura tridimensionale in questione è stata ottenuta da una linea cellulare tumorale. Sfruttando la capacità del microscopio a 2 fotoni di penetrare senza danneggiare il campione, lo sferoide è stato visualizzato a più livelli di profondità in live-imaging. In figura è possibile notare l’immagine di una porzione centrale dello sferoide, nella quale è stato determinato l’indice metabolico, riscontrando un netto shift verso il metabolismo ossidativo.
Conclusione
L’utilizzo dell’imaging olotomografico di Nanolive e dell’analisi dei fasori di FLIM LABS rappresenta una soluzione avanzata e non invasiva per lo studio del metabolismo cellulare in real-time. Queste strategie permettono di monitorare dinamicamente i cambiamenti metabolici senza l’impiego di marcatori esogeni, preservando la fisiologia cellulare. L’approccio label-free consente una quantificazione oggettiva, ripetibile e ad alta risoluzione delle variazioni fenotipiche legate a stress, differenziamento o trattamenti farmacologici. Tali tecnologie offrono un vantaggio competitivo per laboratori di ricerca, aziende biotech e farmaceutiche impegnate nello sviluppo di terapie personalizzate, drug screening e analisi funzionali. L’utilizzo di questi approcci apre nuove prospettive nell’analisi metabolica, accelerando processo di ricerca e sviluppo.


