Negli ultimi anni l’olotomografia di Nanolive è diventata sempre più conosciuta grazie ai numerosi e suggestivi video pubblicati su riviste scientifiche e sui social. A chi non ha avuto l’occasione di apprezzare i video prodotti con gli stumenti Nanolive consigliamo vivamente una visita al loro sito web (Nanolive): basteranno pochi secondi per innamorarsi di Nanolive. La risoluzione subcellulare, la fluidità del video, i dettagli dei complessi movimenti cellulari; gli esperimenti risultano esteticamente affascinanti, ma anche completi e informativi. Infatti, l’occhio vuole la sua parte, ma è altrettanto vero che il ricercatore vuole risultati. E dunque come possiamo tradurre i suggestivi e complessi video Nanolive in risultati concreti? La risposta è EVE Analytics (EA), il software d’analisi associato ai microscopi Nanolive (Figura 1).
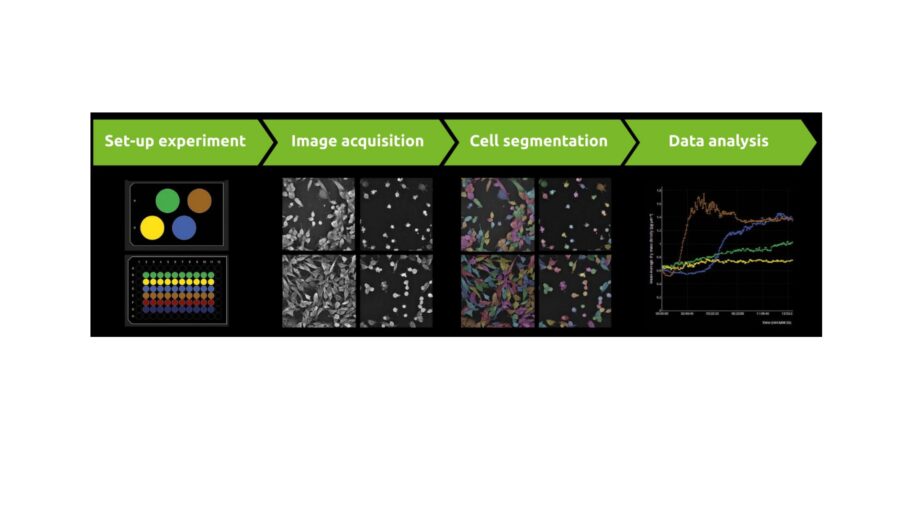
EVE Analytics è un programma ottimizzato per i video Nanolive che effettua una precisa segmentazione delle cellule e fornisce 13 parametri descriventi cell content, cell morphology e cell population. EA massimizza la rilevanza biologica di ogni esperimento estraendo dati unbiased e multiplexed per ogni frame; oltre a aumentare la robustezza dei risultati ottenuti, la precisa e continua analisi del software aumenta la possibilità di rilevare effetti transienti che verrebbero persi con end-point assay. L’interfaccia user-friendly di EA rende le analisi Nanolive veloci e ottimizzate al fine di ottenere il massimo dei risultati nel minor tempo possibile. È infatti sufficiente un semplice click al termine dell’esperimento per attivare l’analisi. L’analisi è semi-automatica in quanto è necessario l’intervento dell’operatore con qualche semplice click. Innanzitutto si dovrà selezionare nome e tipo di analisi [1] e poi seguiranno due step.
[1] Esistono varie tipologie di analisi EA suddivise in moduli. La versione base include il modulo Standard, ossia i 13 parametri descritti in questo articolo. Le altre analisi disponibili sono moduli ottimizzati per esperimenti specifici e potete trovare una loro descrizione nei vari articoli di Media System Lab o direttamente sul sito web di Nanolive.

Il primo step si tratta della clusterizzazione delle immagini: EA raggruppa frames simili sulla base della loro distribuzione dell’indice di rifrazione (Figura 2). Questo step permette all’operatore di selezionare solamente i cluster d’interesse e di escludere tutti i pozzetti non significativi (es. contaminazione, morte cellulare, ecc.).
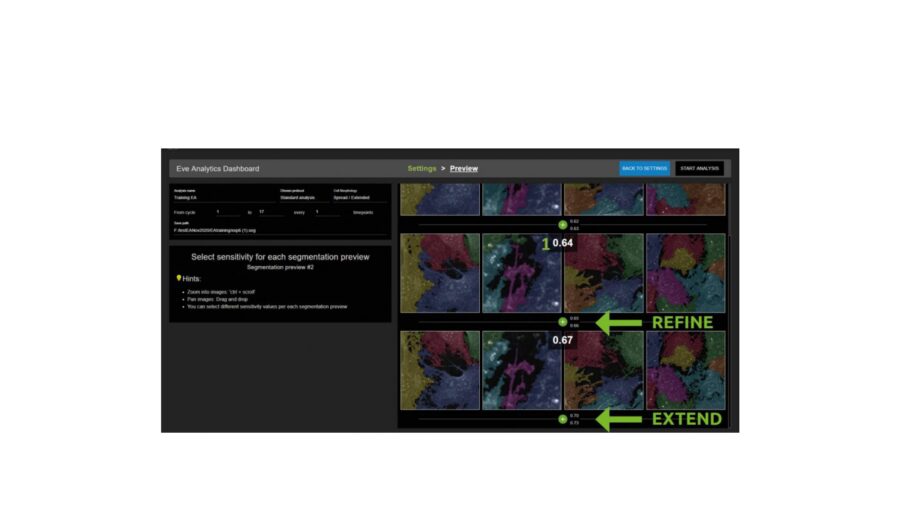
Terminata la clusterizzazione, il secondo step prevede la selezione del parametro di sensibilità (Figura 3). In questa fase, l’operatore potrà definire accuratamente la threshold che meglio segmenta le cellule nelle immagini. In automatico il software mostrerà 3 livelli di sensibilità: nella maggior parte dei casi una delle 3 threshold proposte sarà la migliore, ma nel caso in cui non fossimo soddisfatti potremmo regolare il parametro fino all’ottenimento della segmentazione gradita.
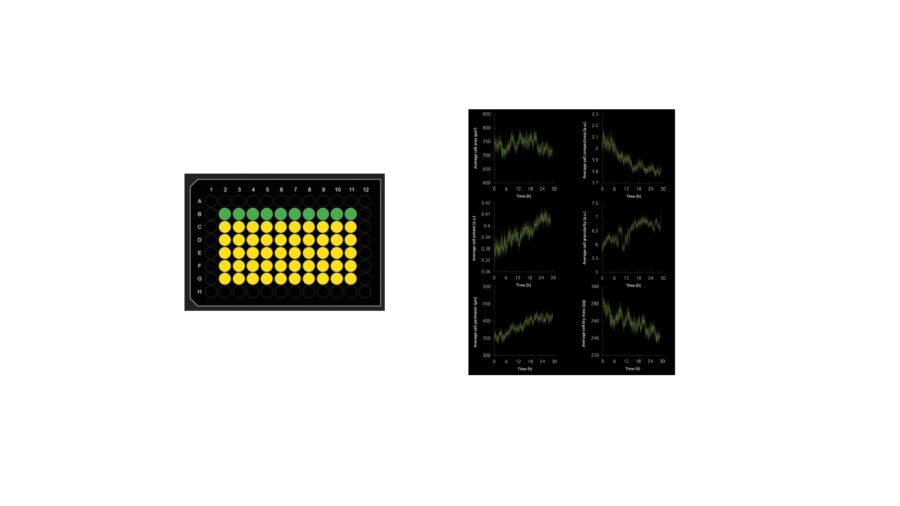
Definita la sensibilità, EA effettuerà l’analisi e otterremo i 13 parametri per tutti i pozzetti dell’esperimento (Figura 4). Raggruppando più pozzetti otterremo direttamente la media dei tali, altrimenti selezionandoli singolarmente otterremo i dati per ognuno. I risultati vengono mostrati in grafici personalizzabili e esportabili (in formato .csv), permettendo all’operatore di visualizzare ogni singolo parametro per ogni gruppo creato.
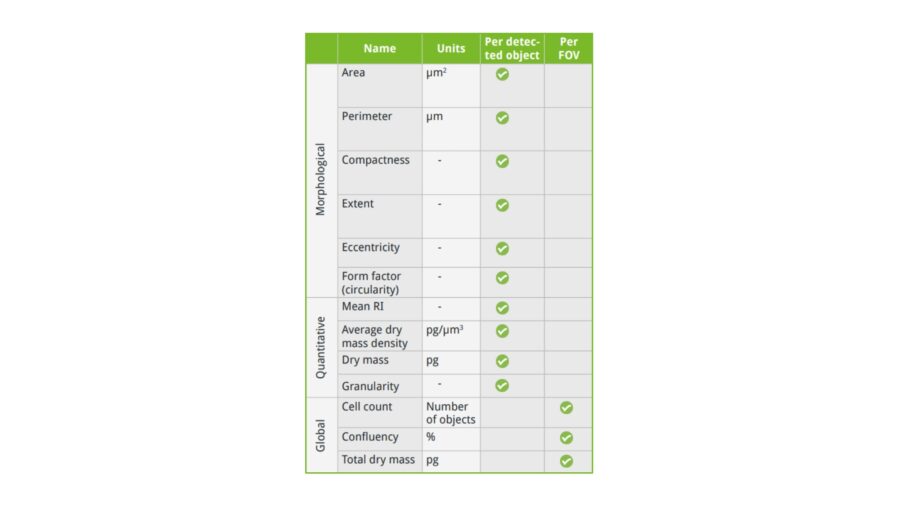
I parametri forniti da EA possono essere suddivisi in 3 categorie: cell content, cell morphology e cell population (Figura 5). I primi 6 parametri elencati riguardano la morfologia della cellula: ogni detected object verrà analizzato per area, perimetro, circolarità, ecc, fornendo così una completa descrizione della morfologia dell’intera coltura cellulare. In fondo alla lista troviamo Cell Count e Confluency, i quali invece forniscono una quantificazione precisa del numero di cellule e della percentuale di area occupata da esse (cell population).
I parametri più intriganti però si basano sull’indice di rifrazione e sono dunque esclusivi dell’olotomografia. Questi parametri sono dry mass e granularity, i quali possono essere quantificati grazie alla tecnologia Nanolive. Nello specifico, con dry mass si intende la massa secca della cellula, dunque tutto il contenuto cellulare esclusa l’acqua (le macromolecole). Il parametro granularity invece si riferisce alla distribuzione della dry mass ed è dunque un indice di distribuzione dei componenti cellulari e degli organelli, molto utile per comprendere lo stato della popolazione in analisi (per esempio situazioni di stress).
Questi due parametri forniscono informazioni precise riguardo la composizione cellulare (cell content), ma anche sulle condizioni generali dell’intera popolazione (cell population). Assieme alle altre 11 sopra elencate, le metrics di EA permettono di ottenere dati immediati dagli esperimenti Nanolive. Sarà sufficiente una breve osservazione dei 13 parametri per comprendere l’andamento dell’esperimento, le differenze tra i vari pozzetti, le condizioni del campione o gli effetti del trattamento. Per un’analisi più dettagliata invece, sarà possibile esportare i raw data in file .csv e valutare ogni singolo punto per ogni pozzetto.
In conclusione, EA è un software ottimizzato per l’analisi di long-term live imaging experiment, facile da utilizzare e in grado di fornire dati robusti e unbiased in qualche semplice click.





