Escluse rarissime eccezioni, in ogni cellula del nostro corpo è presente un nucleo. Il nucleo è il centro di controllo della cellula e al suo interno troviamo il nostro materiale genetico, le istruzioni della vita che determinano il fenotipo della cellula e dell’intero organismo. Tuttavia, la gestione del DNA non è l’unica funzione di questo organello, infatti anche il suo posizionamento e i suoi movimenti contribuiscono a vari eventi cellulari. Posizione e movimenti del nucleo all’interno della cellula sono infatti finemente regolati al fine di specifiche funzioni come migrazione cellulare o mitosi.
Ne è un esempio la rotazione nucleare, la quale fu descritta per la prima volta negli anni 50, ma venne ignorata fino agli anni 80 quando vennero proposti due modelli a spiegazione di tali rotazioni. Nel primo caso si ipotizzava la sola rotazione del materiale intranucleare lasciando immutata la posizione della membrana nucleare, mentre il secondo modello suggeriva una rotazione nucleare totale, inclusa la membrana nucleare e il citoscheletro associato circostante.
Tale quesito è rimasto irrisolto per anni perché considerato irrilevante, ma anche per l’incapacità tecnica di visualizzare dinamiche rapide e complesse con la microscopia convenzionale. Grazie all’evoluzione tecnologica ora disponiamo di microscopi all’avanguardia e di vario genere che ci permettono di affrontare domande biologiche con strategie differenti. Dall’ottica alla fluorescenza, la microscopia ha contribuito enormemente alla descrizione di numerosi processi subcellulari. Tuttavia, alcune limitazioni tecniche come la bassa risoluzione o la fototossicità (per approfondire puoi leggere la application note “Video killed the imaging star) impediscono la visualizzazione dettagliata di dinamiche rapide e nel lungo periodo.
Una soluzione ai limiti della microscopia convenzionale è rappresentata dall’olotomografia, tecnologia innovativa alla base dei microscopi Nanolive. I microscopi Nanolive sono in grado di visualizzare cellule in 3D e in time-lapse, ad alta risoluzione e senza alterare il campione. Nanolive è infatti una tecnologia label-free, dunque non invasiva e senza danni da fototossicità. Tale caratteristica comporta l’enorme vantaggio di poter visualizzare le cellule nel tempo senza limiti di frequenza e durata, e senza introdurre bias nell’esperimento. Infine, l’alta risoluzione spaziale (200nm XY) e temporale (1 immagine ogni 2 secondi) permette di osservare con grande precisione dettagli e dinamiche subcellulari. Grazie a queste caratteristiche, Nanolive ha aperto nuovi orizzonti nel live-cell imaging dando la possibilità di trovare risposta a quesiti rimasti in sospeso, come la rotazione nucleare. Attraverso l’olotomografia, i ricercatori dell’EPFL di Losanna hanno condotto uno studio sui movimenti degli organelli trovando risposta al quesito irrisolto riguardo la rotazione del nucleo [1]. Filmando per ore cellule staminali di topo, Sandoz e colleghi hanno osservato la rotazione nucleare, la quale coinvolge chiaramente lo spinning dei nucleoli e del carioplasma, ma anche della membrana nucleare.
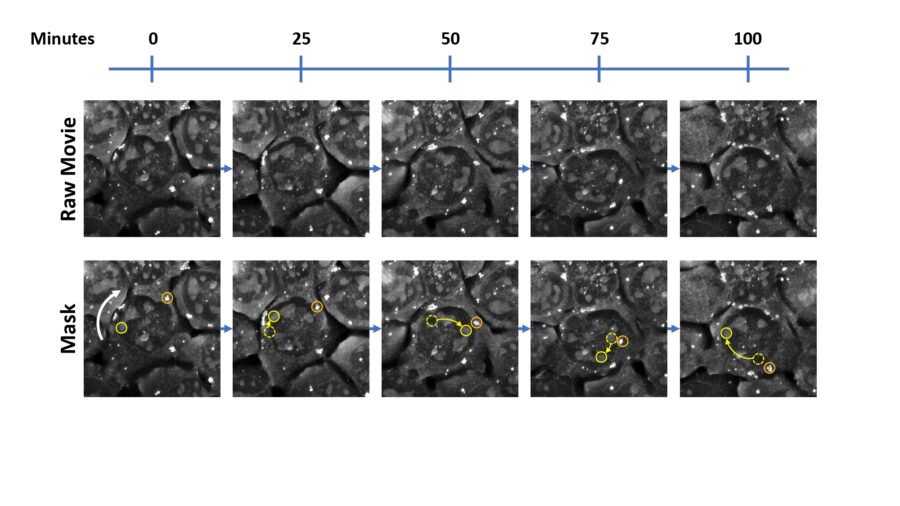
Inoltre, la lenta rotazione delle lipid droplets attorno al nucleo, lascia presupporre un coinvolgimento del citoscheletro circostante con conseguente generazione di uno stream citosolico.
Una dettagliata analisi informatica dei video ha permesso infine di quantificare precisamente tempistiche, entità e velocità del nuclear spinning. Tale evento è risultato molto vario in tutti e tre i parametri. La rotazione nucleare può avvenire in 45 minuti o 10 ore, essere di soli 80 gradi oppure sequenziale fino a 700 gradi, con velocità che vanno dai 0,2 gradi/min-1 ai 13 gradi/min-1.
In conclusione, Nanolive è una tecnologia label-free che permette l’osservazione di numerose dinamiche subcellulari ad alta risoluzione spaziale e temporale. Grazie all’approccio unbiased e multiplexed di Nanolive, è stato possibile estrapolare una risposta completa e dettagliata attraverso la visualizzazione di più organelli simultaneamente (nucleo, nucleoli, membrana nucleare e lipid droplets) in un singolo esperimento. Infine, le tempistiche e velocità di rotazione molto varie dimostrano la sensibilità di Nanolive nell’ottenere informazioni dettagliate sulle dinamiche subcellulari.
Referenze:
[1] Sandoz PA, Tremblay C, van der Goot FG, Frechin M (2019) Image-based analysis of living mammalian cells using label-free 3D refractive index maps reveals new organelle dynamics and dry mass flux. PLoS Biol 17(12): e3000553





